PRML上巻 P6-7
昨日の続き.

図をみると,次数
のケースは関数
の表現としては明らかに不適切であることがわかる.
このような振る舞いは過学習(over-fitting)として知られている.
我々の目標は,新たなデータに対して正確な予測を行える高い汎化性能の達成である.
そこで汎化性能がにどう依存するかを定量的に評価する.
テスト集合に対して,誤差関数を評価する.このとき,
で定義される平均二乗平方根誤差(root-mean-square error, RMS error)を用いると便利なことがある.
で割ることによりサイズの異なるデータ集合を比較できるようになり,平方根を取ることによって
は目的変数
と同じ尺度であることが保証される.
いろいろなに対する訓練集合とテスト集合のRMS誤差のグラフを描いてみる.
ここで,テスト集合は新たに生成した100個のデータ点からなる.
%matplotlib inline import matplotlib.pyplot as plt import japanize_matplotlib import numpy as np import numpy.polynomial.polynomial as poly np.random.seed(42)
# t = sin(2 \pi x) x1 = np.linspace(0, 1, 100) t1 = np.sin(2 * np.pi * x1) # training data set (N=10) # t = sin(2 \pi x) + gaussian noise x2 = np.linspace(0, 1, 10) t2 = np.sin(2 * np.pi * x2) + np.random.normal(0, 0.2, 10) # test data set (N=100) # t = sin(2 \pi x) + gaussian noise t3 = np.sin(2 * np.pi * x1) + np.random.normal(0, 0.2, 100)
RMSE_train = [] RMSE_test = [] for m in range(10): w = np.polyfit(x2, t2, m) y_train = np.poly1d(w)(x2) y_test = np.poly1d(w)(x1) E_train = 0.5 * np.sum(np.square(y_train - t2)) E_test = 0.5 * np.sum(np.square(y_test - t1)) RMSE_train.append(np.sqrt(2 * E_train / t2.size)) RMSE_test.append(np.sqrt(2 * E_test / t1.size))
M = np.linspace(0, 9, 10) p1 = plt.plot(M, RMSE_train, color='blue', marker='o', markerfacecolor='None', markeredgecolor='blue') p2 = plt.plot(M, RMSE_test, color='red', marker='o', markerfacecolor='None', markeredgecolor='red') plt.xlabel('M') plt.ylabel(r'$E_{RMS}$') plt.legend([p1[0], p2[0]], ['訓練', 'テスト'])
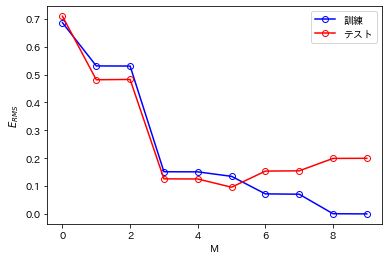
上図をみると,(書籍の図ほど顕著ではないものの)3<Mの範囲で徐々にテスト誤差が大きくなっていることがわかる.
また,では訓練誤差が0になっている.これは多項式の自由度が10であり,訓練集合の10個のデータ点にちょうど当てはめられるからである.
今日はここまで.